
Une nouvelle analyse génomique suggère qu'il existe probablement des dizaines d'espèces inconnues de chauves-souris fer à cheval en Asie du Sud-Est. Ces mammifères, également appelés rhinolophides, sont considérés comme le réservoir de nombreux virus zoonotiques, qui passent des animaux aux humains, y compris les proches parents des virus responsables du syndrome respiratoire aigu sévère (SRAS) et de la COVID-19. L'identification correcte des espèces de chauves-souris pourrait aider à identifier les points chauds géographiques présentant un risque élevé de zoonoses, selon Shi Zhengli, virologue à l'Institut de virologie de Wuhan en Chine. « C'est un travail important », prévient-il.
Pour la co-auteure de l'étude, Alice Hughes, biologiste de la conservation à l'Université de Hong Kong, une meilleure identification des espèces de chauves-souris inconnues pourrait également soutenir la recherche des origines du SARS-CoV-2 en réduisant les lieux de recherche des chauves-souris susceptibles d'abriter des proches parents du virus. Les plus proches parents connus du SARS-CoV-2 ont été trouvés chez des chauves-souris Rhinolophus affinis dans la province du Yunnan, dans le sud-ouest de la Chine, et chez trois espèces de chauves-souris fer à cheval dans Laos.
Espèces cryptiques
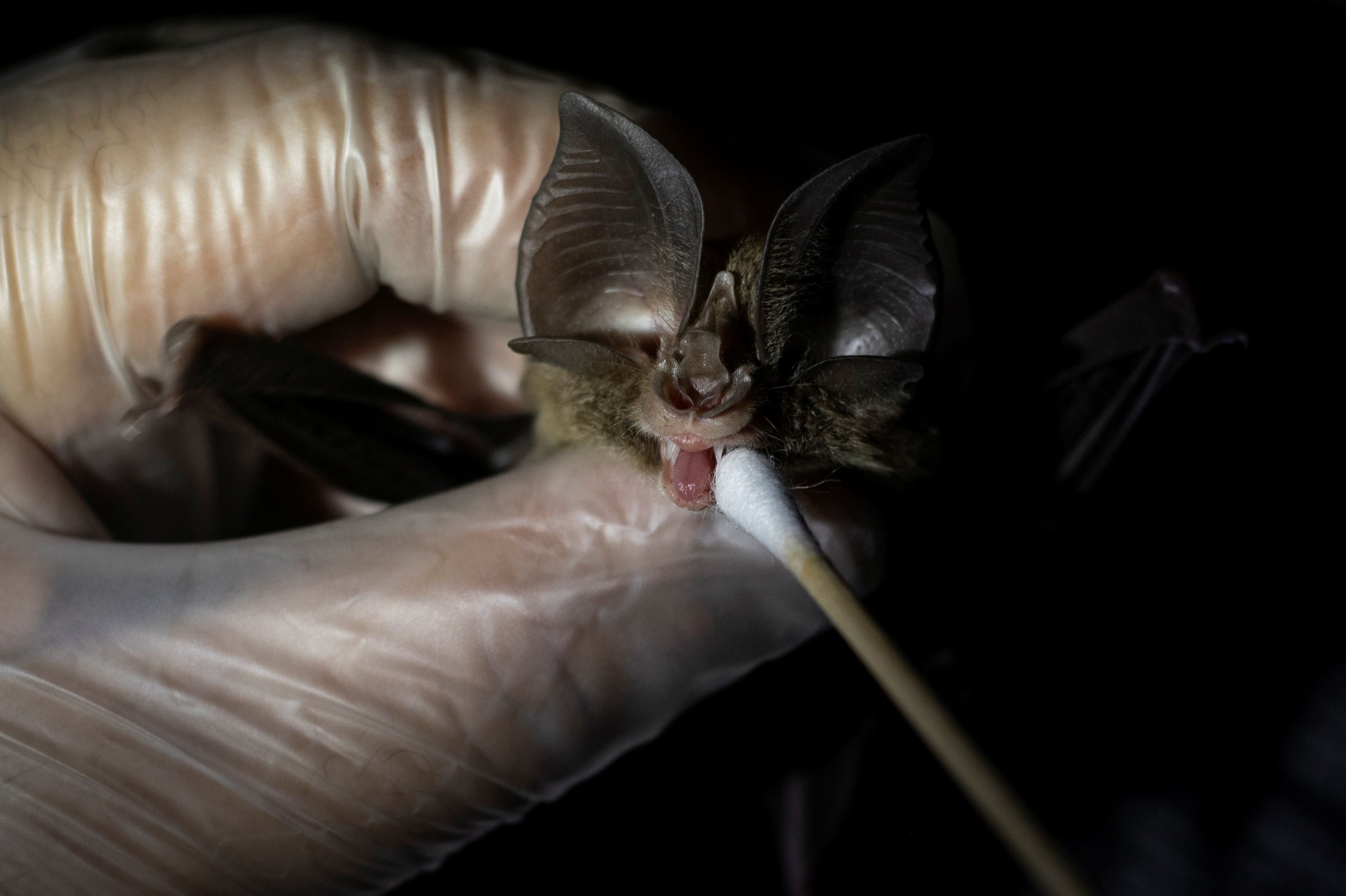
Hughes souhaitait mieux comprendre la diversité des chauves-souris en Asie du Sud-Est et trouver des moyens normalisés pour les identifier. Elle et ses collègues ont donc attrapé des chauves-souris dans le sud de la Chine et en Asie du Sud-Est entre 2015 et 2020. Ils ont pris des mesures et des photographies des ailes et de la lame du nez des chauves-souris, « l'étrange ensemble de tissus autour du nez », tel que décrit par Hughes, et ont enregistré leurs appels à l'écholocation. Ils ont également prélevé une petite quantité de tissu sur les ailes des chauves-souris pour extraire des données génétiques.
Pour cartographier la diversité génétique des chauves-souris, l'équipe a utilisé des séquences d'ADN mitochondrial de 205 de leurs animaux capturés et 655 autres séquences de bases de données en ligne, représentant un total de 11 espèces de rhinolophidés. En règle générale, plus la différence entre les génomes de deux chauves-souris est grande, plus les animaux sont susceptibles de représenter des groupes génétiquement distincts et, par conséquent, des espèces différentes.

Les chercheurs ont découvert que chacune des 11 espèces était probablement en fait plusieurs espèces, y compris peut-être des dizaines d'espèces cachées dans l'échantillon. Les espèces cachées ou « cryptiques » sont des animaux qui semblent appartenir à la même espèce mais qui sont génétiquement différents. Par exemple, la diversité génétique de Rhinolophus sinicus suggère que le groupe pourrait être composé de six espèces distinctes. Dans l'ensemble, ils ont estimé qu'environ 40 % des espèces en Asie n'ont pas été officiellement décrites.
« C'est un chiffre qui donne à réfléchir, mais qui n'est pas vraiment surprenant », explique Nancy Simmons, conservatrice de l'American Museum of Natural History de New York. » Les chauves-souris rhinolophyïdes forment un groupe complexe et il n'y a eu qu'un échantillon limité d'animaux », explique-t-il.
Cependant, pour le spécialiste, le fait de s'appuyer sur l'ADN mitochondrial pourrait signifier que le nombre d'espèces cachées est surestimé. En effet, l'ADN mitochondrial n'est hérité que de la mère, de sorte que des informations génétiques importantes peuvent manquer, explique le spécialiste. Néanmoins, l'étude pourrait mener à une explosion de recherches visant à nommer de nouvelles espèces de chauves-souris dans la région.
Plus de preuves
« Les résultats corroborent d'autres recherches génétiques qui suggèrent qu'il existe de nombreuses espèces cryptiques en Asie du Sud-Est », explique Charles Francis, biologiste au Service canadien de la faune, de l'environnement et des changements climatiques à Ottawa, qui étudie les chauves-souris dans la région. Mais, selon Francis, les estimations sont basées sur un petit nombre d'échantillons.
L'équipe de Hughes a utilisé des données morphologiques et acoustiques pour effectuer une analyse plus détaillée de 190 chauves-souris trouvées dans le sud de la Chine et au Vietnam et a constaté que cela appuyait leur découverte selon laquelle de nombreuses espèces n'avaient pas été identifiées dans ces régions. L'étude avance un argument solide en faveur de « l'utilisation de plusieurs sources de données pour délimiter les espèces », explique M. Simmons.
Hughes souligne que son équipe a également découvert que le lambeau tissulaire juste au-dessus des narines des chauves-souris, appelé selle turque, pouvait être utilisé pour identifier des espèces sans avoir besoin de données génétiques. Pour Gábor Csorba, taxonomiste au Musée hongrois d'histoire naturelle de Budapest, « cela signifie que des espèces cachées pourraient être identifiées sans études morphologiques intrusives ni analyses coûteuses de l'ADN ».
CONTINUEZ À LIRE :
Más Noticias
Debanhi Escobar : ils ont sécurisé le motel où elle a été retrouvée sans vie dans une citerne

La personne la plus âgée du monde est décédée à l'âge de 119 ans

Découverte macabre au CDMX : ils ont laissé un corps ensaché et attaché dans un taxi
Les aigles d'Amérique affronteront Manchester City dans un duel de légendes. Voici les détails

Le FBI a perquisitionné des maisons d'un magnat russe lié à Poutine avant la guerre

